En los últimos años, las técnicas moleculares para el diagnóstico etiológico de la endocarditis infecciosa (EI) han adquirido importancia, sin embargo, no están implantadas en todos los laboratorios. Nuestro objetivo es evaluar la utilidad de la técnica de reacción en cadena de la polimerasa (PCR) de amplio espectro seguida de secuenciación (PCR universal) sobre tejido valvular como parte de la rutina diagnóstica de la EI y comparar los resultados con los métodos clásicos.
MétodosSe incluyeron prospectivamente 52 pacientes con EI definitiva y un grupo control con 57 pacientes sin EI según los criterios de Duke modificados del año 2000. Se realizó la PCR universal, además del estudio microbiológico convencional y un examen anatomopatológico.
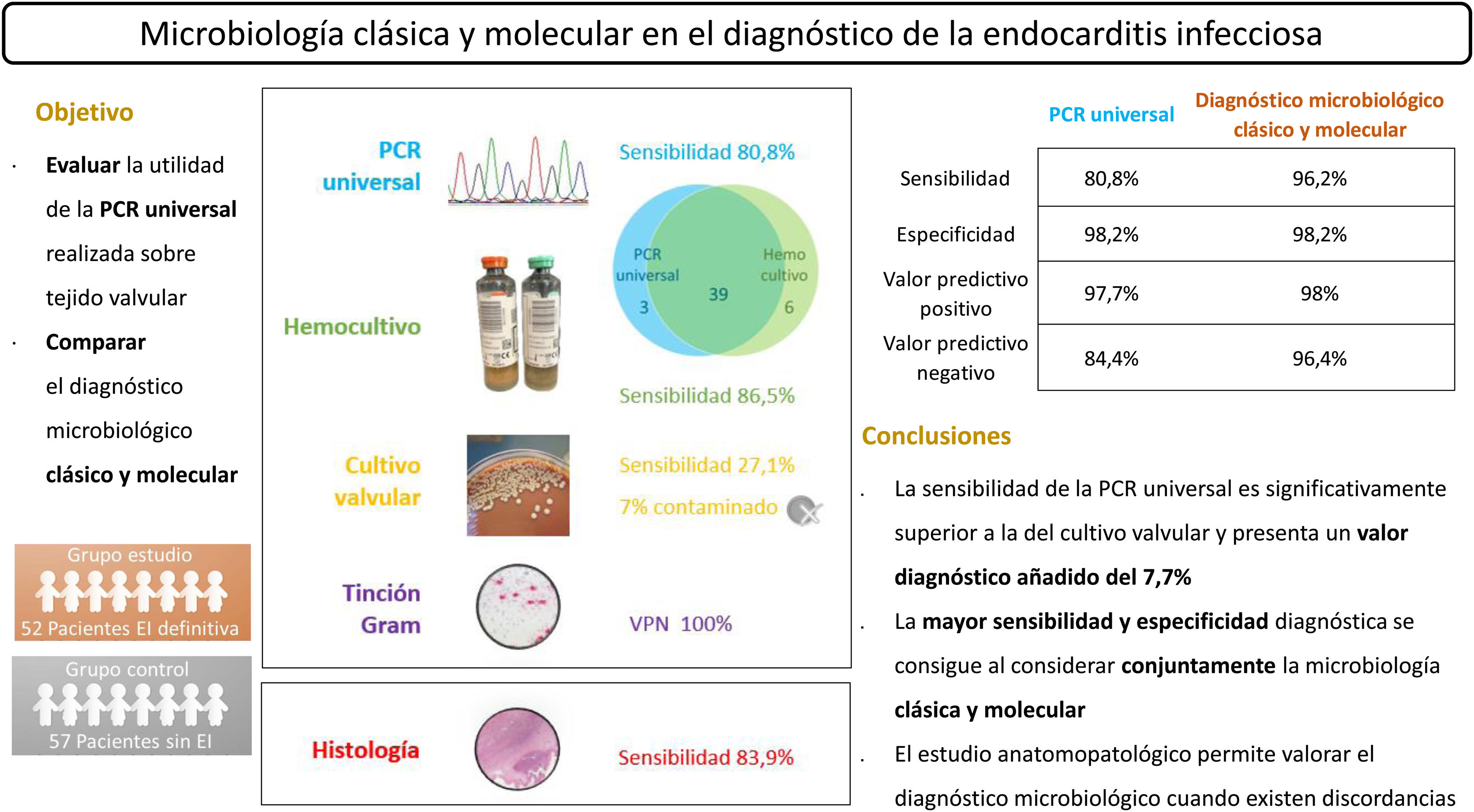
ResultadosLa PCR universal presentó una sensibilidad superior al cultivo valvular (80,8 frente a 27,1%, p<0,001) y permitió identificar el agente causal o confirmarlo en caso de discordancia en el 7,7% de los pacientes. Respecto al diagnóstico convencional, destacan el hemocultivo como técnica de referencia y la tinción de Gram por su elevado valor predictivo positivo. La mayor sensibilidad y especificidad se obtuvo considerando conjuntamente la microbiología clásica y la molecular. El estudio anatomopatológico presentó una sensibilidad del 83,9% y permitió valorar los resultados del diagnóstico microbiológico cuando existieron discordancias.
ConclusionesLa PCR universal demostró ser una técnica valiosa que completa el diagnóstico clásico. En una entidad tan compleja como la EI, es necesario utilizar todas las herramientas diagnósticas a nuestro alcance. Todas las áreas de diagnóstico de laboratorio son complementarias y no excluyentes y deberían ser valoradas conjuntamente.
In recent years, molecular techniques for the aetiological diagnosis of infective endocarditis (IE) have attained relevance; however, they are not implemented in all laboratories. Our objective is to evaluate the usefulness of broad-spectrum polymerase chain reaction (PCR) technique followed by sequencing (universal PCR) on valve tissue as part of the IE diagnostic practice and to compare the results with classical methods.
ResultsUniversal PCR showed a higher sensitivity than valve culture (80.8 vs 27.1%, P<.001) and allowed the identification of the causative agent or the confirmation of the aetiological diagnosis in case of discordance in 7.7% of patients. Regarding conventional diagnosis, blood culture stands out as the reference technique and Gram staining due to its high positive predictive value. The highest sensitivity and specificity was obtained considering classical and molecular microbiology together. The anatomopathological study presented a sensitivity of 83.9% and made it possible to assess the results of the microbiological diagnosis when there were discrepancies.
ConclusionsUniversal PCR proved to be a valuable technique that completes the classical diagnosis. In an entity as complex as IE, it is necessary to use all the diagnostic tools at our disposal. All areas of laboratory diagnosis are complementary and not exclusive and should be assessed together.
La endocarditis infecciosa (EI) es una enfermedad grave y, aunque poco frecuente, su incidencia ha aumentado en Europa en las últimas décadas1. Presenta una mortalidad elevada en la que influyen la dificultad diagnóstica y las limitaciones en su tratamiento2.
El hemocultivo constituye uno de los pilares diagnósticos, sin embargo, puede ser negativo hasta en el 40% de los pacientes debido al tratamiento antibiótico prolongado o a microorganismos de difícil crecimiento, como estreptococos defectivos, bacterias del grupo HACEK, Bartonella spp., Coxiella burnetii o Tropheryma whipplei. Las técnicas moleculares representan una alternativa para la identificación de los agentes causales de EI, siendo especialmente beneficiosas en pacientes con hemocultivo negativo3,4.
La técnica más ampliamente utilizada en pacientes sometidos a cirugía es la secuenciación directa de microorganismos (polymerase chain reaction [PCR, «reacción en cadena de la polimerasa»] universal) a partir de tejido valvular. Consiste en la amplificación y posterior secuenciación de regiones de genes conservados de bacterias (gen 16S ácido ribonucleico ribosomal [ARNr]) y hongos (gen 18S ARNr). La identificación se establece por comparación de la secuencia de ácido desoxirribonucleico (ADN) obtenida con secuencias conocidas depositadas en bases de datos5. Se utilizó por primera vez exitosamente en 19976, y en 2001 se propuso su inclusión en los criterios de Duke modificados de 20007,8, aunque no ha sido incorporada hasta 20239. Cabe señalar que son procedimientos complejos, no estandarizados, y se ha descrito variabilidad en los resultados publicados10.
El objetivo principal del presente estudio es comprobar la validez de la técnica de PCR universal a partir de tejido valvular en nuestro laboratorio. Otros objetivos son investigar la contribución de las técnicas moleculares al diagnóstico etiológico, analizar la relación entre la microbiología molecular y la clásica, y comparar los resultados del diagnóstico microbiológico y del estudio histológico del tejido valvular.
MétodosCaracterísticas del estudio y pacientesEstudio observacional, prospectivo, comparativo de los pacientes diagnosticados de endocarditis e intervenidos quirúrgicamente entre 2017 y 2019 en un hospital terciario público que atiende a 300.000 habitantes y es centro de referencia de cirugía cardiaca.
El grupo de estudio lo formaron pacientes mayores de 18 años diagnosticados de EI definitiva o posible, según los criterios de Duke modificados de 2000, intervenidos para resecar la válvula infectada. El grupo control incluyó pacientes mayores de 18 años sometidos a cirugía electiva por disfunción valvular sin sospecha de EI diagnosticados según protocolos establecidos con ecocardiografía Doppler. También se incluyeron en este grupo pacientes con sospecha inicial de EI en los que posteriormente se descartó la infección.
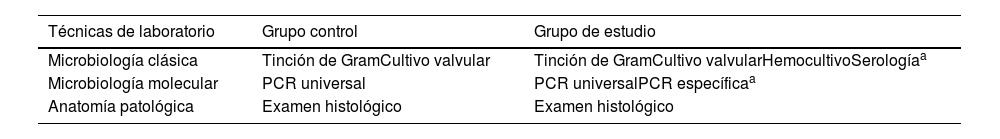
Las técnicas de laboratorio empleadas para cada grupo están reflejadas en la tabla 1. También se recopilaron datos clínicos, demográficos, ecocardiográficos y quirúrgicos, factores de riesgo, antecedentes de EI y duración del tratamiento antimicrobiano en el momento de la cirugía.
Técnicas de laboratorio realizadas en los pacientes del grupo de estudio y del grupo control
| Técnicas de laboratorio | Grupo control | Grupo de estudio |
|---|---|---|
| Microbiología clásica | Tinción de GramCultivo valvular | Tinción de GramCultivo valvularHemocultivoSerologíaa |
| Microbiología molecular | PCR universal | PCR universalPCR específicaa |
| Anatomía patológica | Examen histológico | Examen histológico |
PCR: reacción en cadena de la polimerasa.
El estudio fue aprobado por el Comité de Ética del hospital (registro 2017/0271).
Diagnóstico microbiológico clásicoSe llevó a cabo de forma rutinaria en el servicio de microbiología por procedimientos descritos previamente11. Los hemocultivos se incubaron en el sistema VIRTUO (BioMerieux, Francia). Los microorganismos aislados se identificaron por espectrometría de masas en el equipo Vitek MS (BioMerieux, Francia). Las pruebas de sensibilidad se realizaron con paneles de MicroScan (Dade Behring, Estados Unidos) y procedimientos microbiológicos convencionales.
Se realizó la serología de Coxiella burnetii QFeverIFAIgG (FocusDiagnostics, Estados Unidos), Bartonella spp. BartonellaIFAIgG/IFAIgM (FocusDiagnostic, Estados Unidos) y Legionella pneumophila LegionellapneumophilaIgG/IgM (TrinityBiotechCaptia, Estados Unidos).
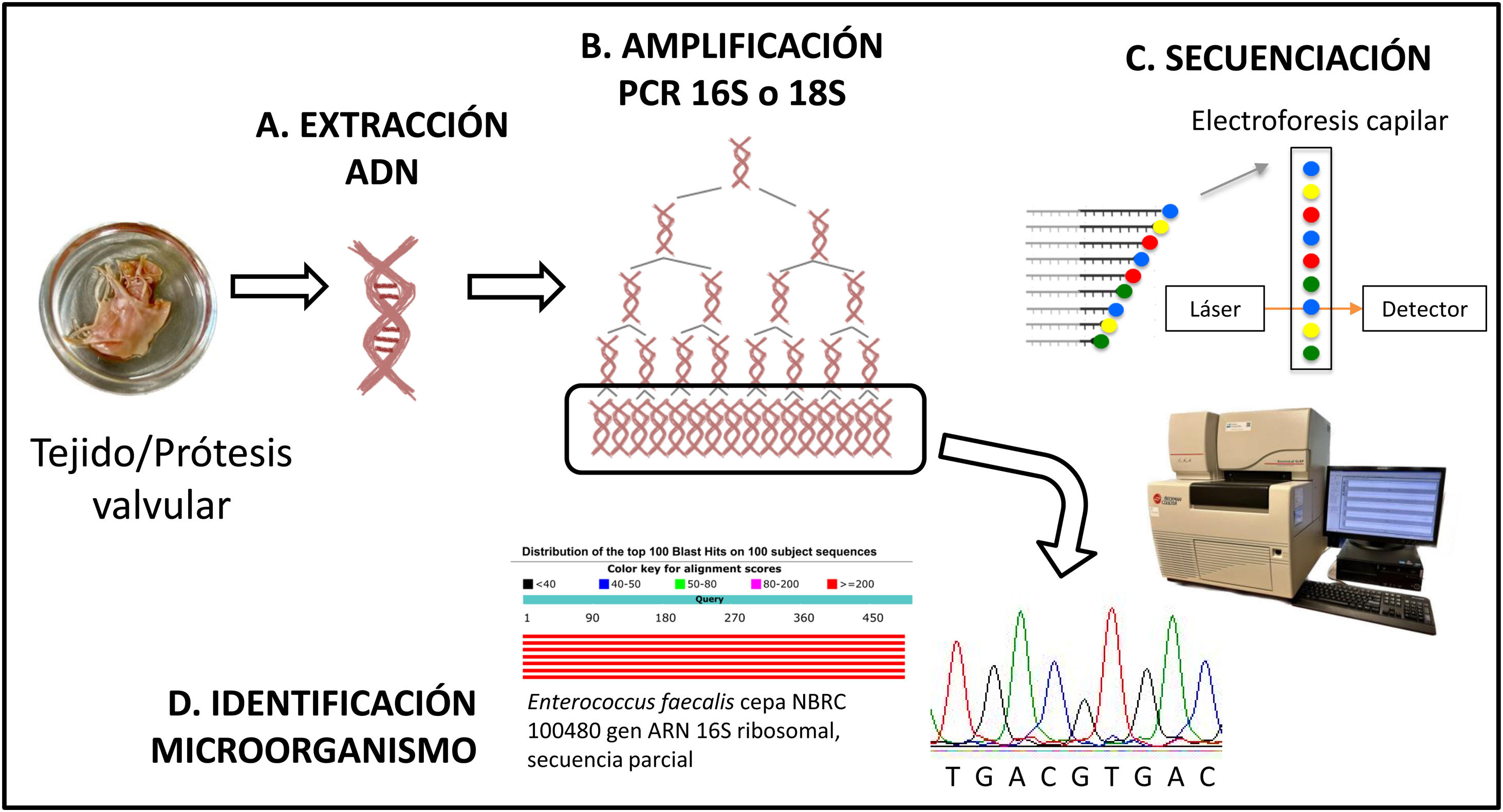
Diagnóstico microbiológico molecularLa figura 1 esquematiza el flujo de trabajo de la PCR de amplio espectro seguida de secuenciación (PCR universal):
- a)
Las muestras de tejido, prótesis, cables o dispositivos se procesaron según procedimientos descritos previamente12 y se realizó la extracción automatizada del ADN en el equipo QiaSymphony (QIAGEN, Países Bajos).
- b)
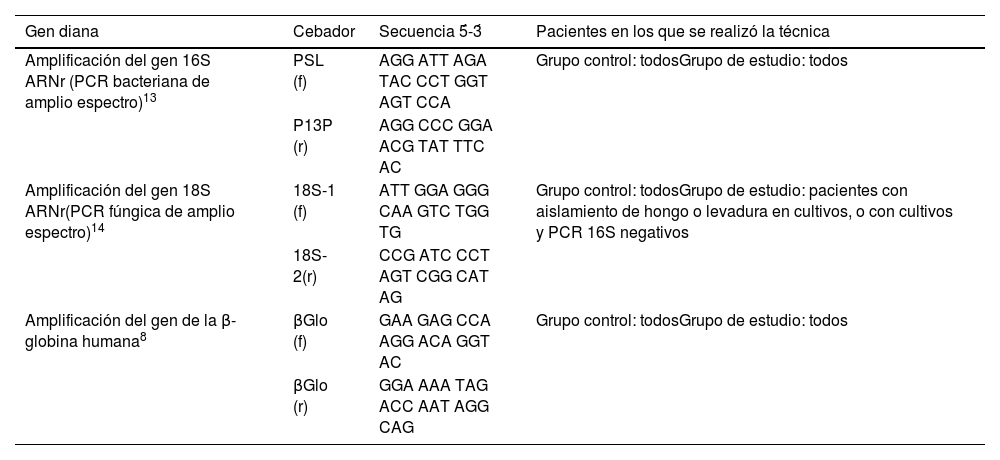
La PCR de amplificación (16S y 18S)12 se realizó en un termociclador Veriti Thermal Cycler (ThermoFisicScientific, Estados Unidos). Los cebadores empleados y en qué pacientes se realizó la técnica se describen en la tabla 2. El producto amplificado se detectó mediante electroforesis en gel de agarosa12.
Tabla 2.Genes diana amplificados y pacientes en los que se realizó la técnica
Gen diana Cebador Secuencia 5́-3́ Pacientes en los que se realizó la técnica Amplificación del gen 16S ARNr (PCR bacteriana de amplio espectro)13 PSL (f) AGG ATT AGA TAC CCT GGT AGT CCA Grupo control: todosGrupo de estudio: todos P13P (r) AGG CCC GGA ACG TAT TTC AC Amplificación del gen 18S ARNr(PCR fúngica de amplio espectro)14 18S-1 (f) ATT GGA GGG CAA GTC TGG TG Grupo control: todosGrupo de estudio: pacientes con aislamiento de hongo o levadura en cultivos, o con cultivos y PCR 16S negativos 18S-2(r) CCG ATC CCT AGT CGG CAT AG Amplificación del gen de la β-globina humana8 βGlo (f) GAA GAG CCA AGG ACA GGT AC Grupo control: todosGrupo de estudio: todos βGlo (r) GGA AAA TAG ACC AAT AGG CAG ARNr: ácido ribonucleico ribosomal; PCR: reacción en cadena de la polimerasa.
- c)
Para la PCR de secuenciación se empleó el kit GenomeLab Dye Terminator Cycle Sequencing with Quick Start (BeckmanCoulter, Estados Unidos) utilizando los mismos cebadores que en la PCR de amplificación12, y la electroforesis capilar se realizó en el equipo GenomeLab™ (GeneticAnalysisSystem, Estados Unidos).
- d)
Las secuencias obtenidas se cotejaron en la web de GenBank con el software BLAST15. Los microorganismos se identificaron según criterios del Clinical and Laboratory Standards Institute16.
Se realizó la PCR específica en tejido valvular de Coxiella burnetii17, Bartonella spp.18 o Tropheryma whipplei19.
Análisis de datosPara el cálculo de los parámetros estadísticos de la PCR universal se valoraron conjuntamente las técnicas de PCR 16S y 18S. Para el diagnóstico microbiológico clásico se consideraron los resultados del hemocultivo, el cultivo valvular y la serología. Para el diagnóstico microbiológico global se consideraron los resultados del hemocultivo, el cultivo valvular, la serología, la PCR universal y la PCR específica.
Los cultivos de tejido valvular con algún aislamiento en pacientes sin EI se catalogaron como contaminados, apoyados por el resultado negativo de las técnicas moleculares.
Para el cálculo de sensibilidad, especificidad, valor predictivo positivo (VPP), valor predictivo negativo (VPN) e intervalos de confianza (IC), considerando los criterios de Duke modificados de 2007 como patrón de referencia, se utilizó la web VassarStats20.
El análisis comparativo se realizó mediante el programa SPSS 12.5, considerándose significativo un valor de p<0,05.
ResultadosEl grupo de estudio incluyó 52 pacientes con EI definitiva y 2 con EI posible. El grupo control sumó 57 pacientes, 42 pacientes sin EI sometidos a cirugía electiva por disfunción valvular y 15 pacientes con sospecha inicial de EI en los que se descartó la infección.
Pacientes con endocarditis infecciosa definitivaLa edad media fue de 62,1 (48,7-75,5) años, 34 varones y 18 mujeres. En 33 casos (63,5%) estaba afectada la válvula nativa (12 aórtica, 11 mitral, 5 aorticomitral, 2 tricúspide, una pulmonar y 2 tricúspide-mitral), en 15 (28,8%) la válvula protésica (11 aórticas, 3 mitrales y una aorticomitrales), 2 casos (3,8%) fueron sobre dispositivo de estimulación cardiaca y hubo otros 2 casos (3,8%) mixtos: sobre válvula nativa y protésica, y sobre válvula protésica y dispositivo de estimulación cardiaca.
Se procesaron 83 especímenes de los 52 pacientes. En 29 pacientes (55,8%) se envió una sola muestra al laboratorio, en 18 (34,6%) se remitieron 2 muestras y en 5 (9,6%) se recogieron 3 o más muestras.
La duración del tratamiento antibiótico en el momento de la cirugía en 38 pacientes fue superior a 5 días, en un paciente 3 días y en 13 pacientes de 24-48h.
Se detectó el agente causal mediante PCR universal (16S y 18S) en 42 pacientes (tabla 3).
Microorganismos identificados por PCR universal y por hemocultivo
| PCR universal positiva y hemocultivo positivo39 de 52 | PCR universal positiva y hemocultivo negativo3 de 52 | PCR universal negativa y hemocultivo positivo6 de 52 |
|---|---|---|
| Staphylococcus aureus (13)Staphylococcus capitis (1)Staphylococcus epidermidis (7)Enterococcus faecalis (4)Granulicatella spp. (1)Streptococcus spp. (5)Streptococcus anginosus (1)Streptococcus gallolyticus (2)Streptococcus mitis/oralis (2)Streptococcus pneumoniae (1)Candida auris (1)Candida parapsilosis (1) | Fusarium spp. (1)Haemophilus parainfluenzae (1)Streptococcus gallolyticus (1) | Staphylococcus aureus (3)Staphylococcus cohnii (1)Streptococcus pneumoniae (1)Streptococcus sanguinis (1) |
Los datos expresan n.
PCR: reacción en cadena de la polimerasa.
En 2 pacientes con EI fúngica, identificada mediante la PCR 18S, la PCR 16S también resultó positiva para Massilia suwonensis y Streptococcus spp.
En 9 pacientes en los que se remitió más de una muestra al laboratorio, la PCR universal fue positiva solo en una de ellas.
Un paciente fue diagnosticado mediante PCR específica de EI por Tropheryma whipplei.
La tinción de Gram presentó un VPN del 100% (IC95% 74,7-100) y una sensibilidad del 36,6% (IC95% 22,6-53,1).
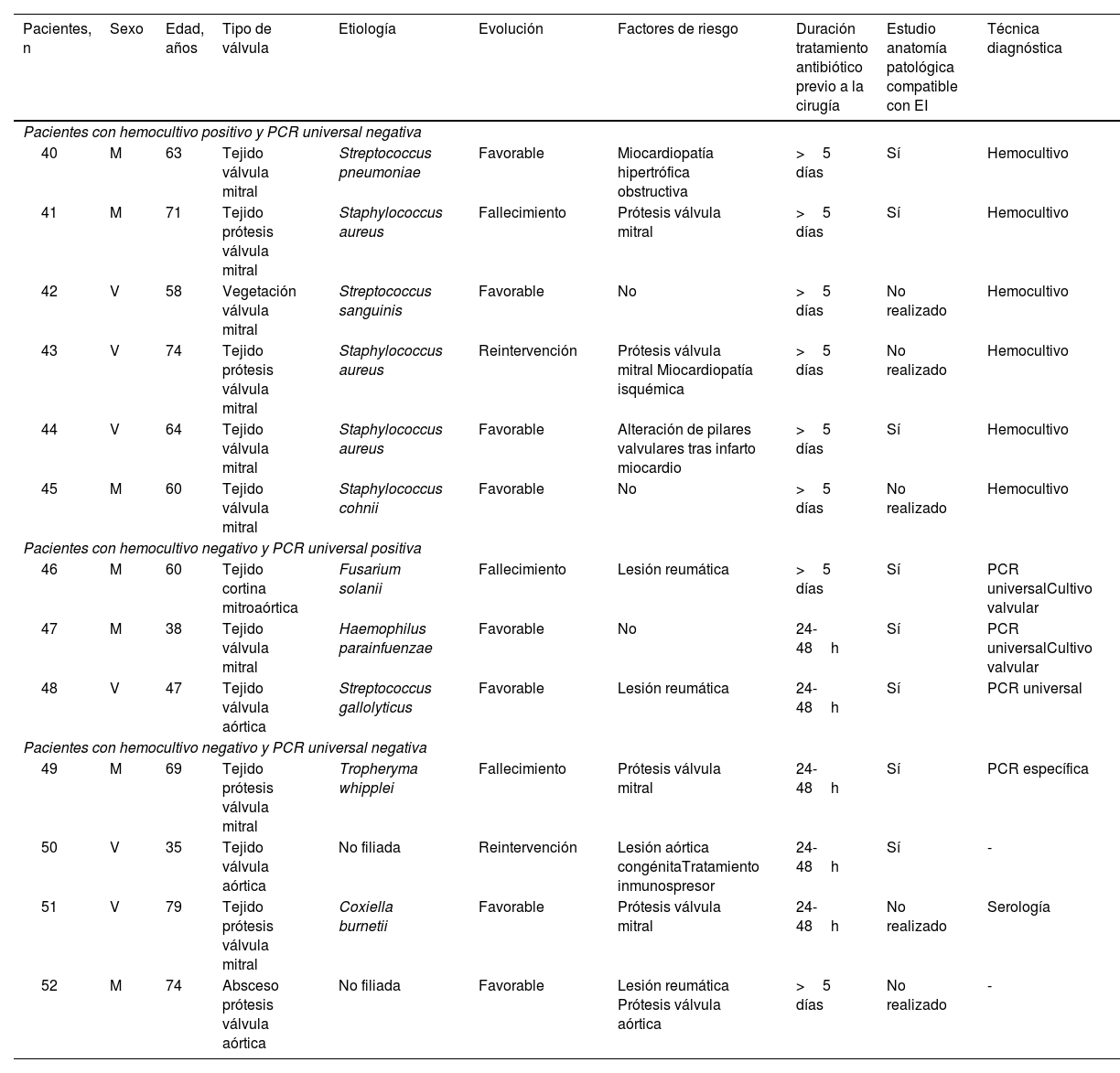
El hemocultivo presentó una sensibilidad del 86,5% (IC95% 73,6-94); los microorganismos identificados se muestran en la tabla 3. La tabla 4 recoge el agente causal y la duración del tratamiento antibiótico en los 7 pacientes (13,5%) en los que el hemocultivo fue negativo.
Características clínicas de los pacientes con hemocultivo positivo en los que la PCR fue negativa y de los pacientes con hemocultivo negativo
| Pacientes, n | Sexo | Edad, años | Tipo de válvula | Etiología | Evolución | Factores de riesgo | Duración tratamiento antibiótico previo a la cirugía | Estudio anatomía patológica compatible con EI | Técnica diagnóstica |
|---|---|---|---|---|---|---|---|---|---|
| Pacientes con hemocultivo positivo y PCR universal negativa | |||||||||
| 40 | M | 63 | Tejido válvula mitral | Streptococcus pneumoniae | Favorable | Miocardiopatía hipertrófica obstructiva | >5 días | Sí | Hemocultivo |
| 41 | M | 71 | Tejido prótesis válvula mitral | Staphylococcus aureus | Fallecimiento | Prótesis válvula mitral | >5 días | Sí | Hemocultivo |
| 42 | V | 58 | Vegetación válvula mitral | Streptococcus sanguinis | Favorable | No | >5 días | No realizado | Hemocultivo |
| 43 | V | 74 | Tejido prótesis válvula mitral | Staphylococcus aureus | Reintervención | Prótesis válvula mitral Miocardiopatía isquémica | >5 días | No realizado | Hemocultivo |
| 44 | V | 64 | Tejido válvula mitral | Staphylococcus aureus | Favorable | Alteración de pilares valvulares tras infarto miocardio | >5 días | Sí | Hemocultivo |
| 45 | M | 60 | Tejido válvula mitral | Staphylococcus cohnii | Favorable | No | >5 días | No realizado | Hemocultivo |
| Pacientes con hemocultivo negativo y PCR universal positiva | |||||||||
| 46 | M | 60 | Tejido cortina mitroaórtica | Fusarium solanii | Fallecimiento | Lesión reumática | >5 días | Sí | PCR universalCultivo valvular |
| 47 | M | 38 | Tejido válvula mitral | Haemophilus parainfuenzae | Favorable | No | 24-48h | Sí | PCR universalCultivo valvular |
| 48 | V | 47 | Tejido válvula aórtica | Streptococcus gallolyticus | Favorable | Lesión reumática | 24-48h | Sí | PCR universal |
| Pacientes con hemocultivo negativo y PCR universal negativa | |||||||||
| 49 | M | 69 | Tejido prótesis válvula mitral | Tropheryma whipplei | Fallecimiento | Prótesis válvula mitral | 24-48h | Sí | PCR específica |
| 50 | V | 35 | Tejido válvula aórtica | No filiada | Reintervención | Lesión aórtica congénitaTratamiento inmunospresor | 24-48h | Sí | - |
| 51 | V | 79 | Tejido prótesis válvula mitral | Coxiella burnetii | Favorable | Prótesis válvula mitral | 24-48h | No realizado | Serología |
| 52 | M | 74 | Absceso prótesis válvula aórtica | No filiada | Favorable | Lesión reumática Prótesis válvula aórtica | >5 días | No realizado | - |
EI: endocarditis infecciosa; M: mujeres; PCR: reacción en cadena de la polimerasa; V: varones.
El cultivo valvular obtuvo una sensibilidad del 27,1% (IC95% 15,7-42,1) y un porcentaje de contaminaciones del 7%. No aportó ninguna identificación que no se hubiera conseguido por PCR universal (fig. 2).
Un paciente fue diagnosticado por serología de EI por Coxiella burnetii.
En 4 pacientes con EI definitiva (7,7%) la PCR universal añadió valor diagnóstico; fue la única técnica capaz de proporcionar el diagnóstico etiológico en un (2%) paciente, confirmó el resultado del cultivo valvular en 2 pacientes con hemocultivo negativo y corroboró el resultado del hemocultivo en un paciente con cultivo valvular contaminado.
El examen histológico del tejido valvular se realizó en 31 de los 52 pacientes, observándose lesiones compatibles con EI en 26 (83,9%) (fig. 2).
Pacientes con endocarditis infecciosa posibleEn uno de los 2 pacientes con EI posible se aisló Staphylococcus hominis en 2 sets de hemocultivo, pero la PCR universal y el cultivo valvular fueron negativos. En el otro paciente no se realizó hemocultivo; en la tinción de Gram del tejido valvular se observaron cocos grampositivos y mediante PCR universal y cultivo valvular se identificó Streptococcus anginosus. En ninguno se realizó estudio anatomopatológico.
Grupo controlLa edad media del grupo control fue de 69,47 (58,68-80,26) años, 38 varones y 19 mujeres. Se retiró la válvula nativa en 53 casos (97%), que fue la válvula aórtica en 27 pacientes (47,4%), la mitral en 27 (47,4%) y ambas en 3 (5,2%).
La PCR 18S resultó negativa en todos los casos. Mediante la técnica 16S se detectó Streptococcus spp. en 3 pacientes (2 de ellos presentaban antecedentes de EI, por lo que no se consideraron para los cálculos de sensibilidad y especificidad).
El cultivo valvular resultó contaminado en el 12% de los casos. En ninguna muestra se observaron microorganismos en la tinción de Gram.
El estudio anatomopatológico se realizó en 48 de los 57 pacientes y fue negativo excepto en 3 casos en los que se identificaron células inflamatorias.
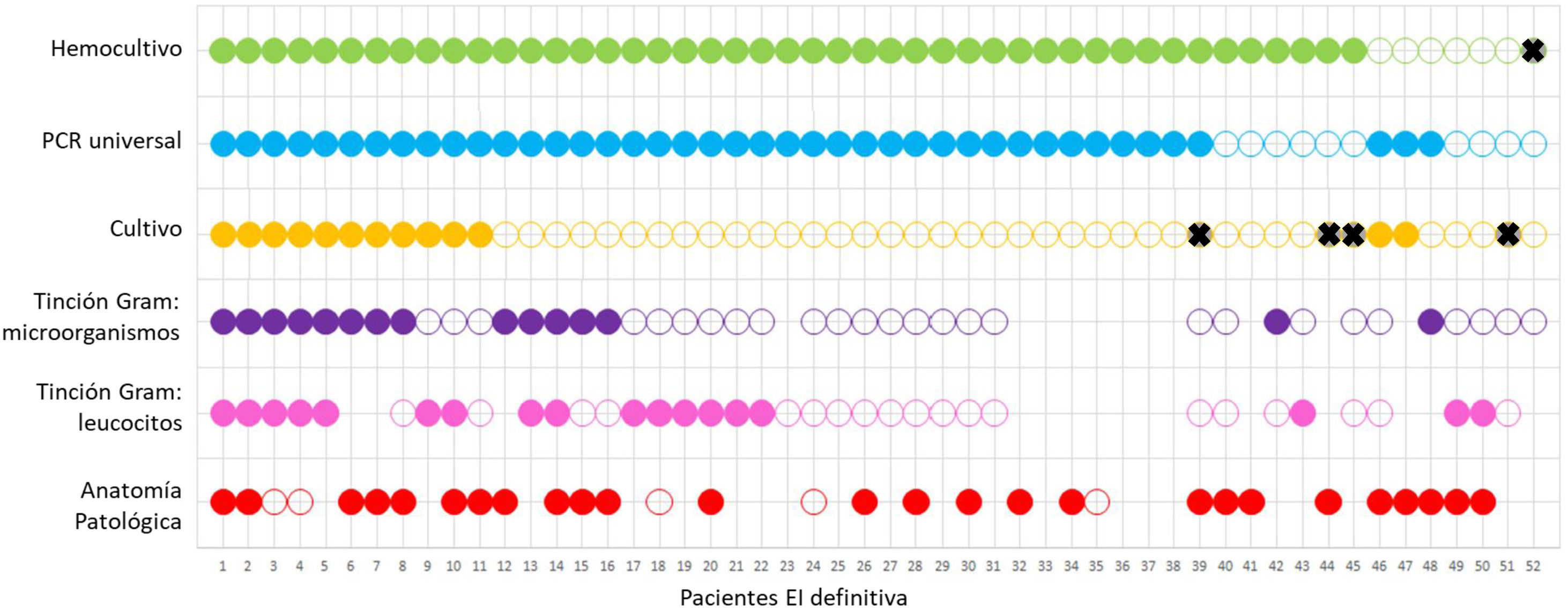
Concordancia y rendimiento diagnósticoEn 39 casos coincidieron el microorganismo identificado mediante hemocultivo y PCR universal, en 6 casos el hemocultivo fue positivo y la PCR universal negativa y en 3 casos el hemocultivo fue negativo, pero mediante PCR se detectó el agente causal (tabla 3). Las características clínicas de los pacientes en los que existieron discrepancias entre hemocultivo y PCR universal y de los 4 pacientes en los que ambas técnicas resultaron negativas se muestran en la tabla 4.
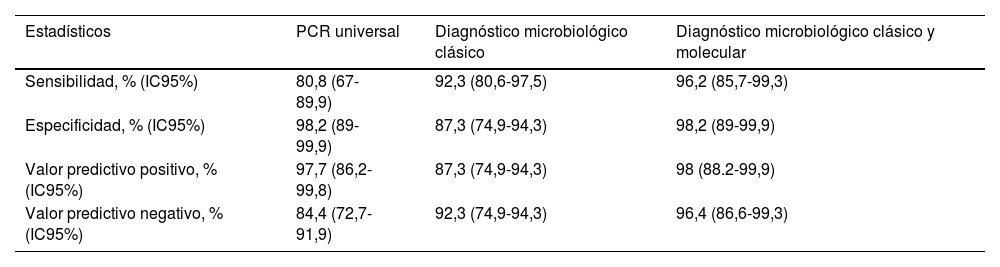
El rendimiento diagnóstico de las distintas técnicas microbiológicas se refleja en la tabla 5. La sensibilidad de la PCR universal (80,8%) y del hemocultivo (86,5%) fueron significativamente superiores a la del cultivo valvular (27,1%), p<0,001.
Rendimiento diagnóstico de las distintas técnicas microbiológicas
| Estadísticos | PCR universal | Diagnóstico microbiológico clásico | Diagnóstico microbiológico clásico y molecular |
|---|---|---|---|
| Sensibilidad, % (IC95%) | 80,8 (67-89,9) | 92,3 (80,6-97,5) | 96,2 (85,7-99,3) |
| Especificidad, % (IC95%) | 98,2 (89-99,9) | 87,3 (74,9-94,3) | 98,2 (89-99,9) |
| Valor predictivo positivo, % (IC95%) | 97,7 (86,2-99,8) | 87,3 (74,9-94,3) | 98 (88.2-99,9) |
| Valor predictivo negativo, % (IC95%) | 84,4 (72,7-91,9) | 92,3 (74,9-94,3) | 96,4 (86,6-99,3) |
IC95%: intervalo de confianza del 95%; PCR: reacción en cadena de la polimerasa.
Se detectó una mayor sensibilidad de la PCR cuando se observaron microorganismos en la tinción de Gram: 14 de 15 pacientes (93,3%) frente a 19 de 26 pacientes (73,1%), aunque no fue estadísticamente significativa (p=0,2).
En relación con la duración del tratamiento antibiótico previo a la cirugía, el porcentaje de cultivos positivos sí que fue significativamente superior en los pacientes con tratamiento antibiótico inferior a 5 días (50 frente a 15,8%, p<0,05), pero esta diferencia no se manifestó en los resultados positivos de la PCR universal.
Por último, no se detectaron diferencias significativas en la sensibilidad de la PCR universal ni del cultivo respecto al sexo, al número de muestras procesadas ni a la positividad del estudio histológico.
DiscusiónLa principal aportación del presente estudio es que la PCR universal mostró una buena sensibilidad, permitiendo determinar la etiología de la EI en el 80,8% de los casos y siendo la única técnica capaz de identificar el agente causal en un paciente. Su realización en más de una muestra aumentó la sensibilidad y podría ser especialmente útil en pacientes en los que se desconoce el agente etiológico en el momento de la cirugía.
Si bien la PCR universal obtuvo buenos resultados de sensibilidad, especificidad, VPP y VPN, otras series presentan mejores parámetros, como la de Mularoni et al., que realizan un enriquecimiento previo de la muestra. Estudios anteriores, en cambio, describen sensibilidades próximas al 50%21,22, aunque con una especificidad del 100%.
En nuestra serie obtuvimos resultados positivos no esperados de la PCR 16S en 2 pacientes con EI fúngica en los que también se detectó Streptococcus spp., que forma parte de la microbiota orofaríngea, y Massilia suwonensis, que es una bacteria ambiental. Las técnicas moleculares amplifican pequeñas cantidades de material genético y son muy susceptibles de contaminación, por lo que en el laboratorio se debe seguir un estricto flujo de trabajo23 que respetamos en todo momento. Respecto al grupo control, se detectó Streptococcus spp. en 3 pacientes, 2 de los cuales presentaban antecedentes de EI por Streptococcus. Se ha descrito que el ADN bacteriano puede persistir detectable en tejidos durante periodos prolongados, incluso años24. En el tercer caso, se podría plantear la posibilidad de una EI pasada no conocida o un falso positivo de la técnica. En una serie similar a la nuestra25 también describen un falso positivo de la PCR 16S en un paciente del grupo control.
En cuanto a la PCR 18S, algunos autores refieren porcentajes importantes de falsos positivos26 indicando que tampoco aportó una identificación que no se hubiera conseguido por cultivo y no recomiendan su realización. En nuestra serie, la PCR 18S no presentó falsos positivos y defendemos su realización porque detecta microorganismos que raramente producen EI y su identificación por diferentes técnicas permite afianzar el diagnóstico.
Excepto algunos trabajos27,28, la mayoría de las publicaciones no hacen alusión al número de muestras procesadas. Nosotros no encontramos diferencias significativas respecto a los resultados positivos de PCR universal, pero 9 pacientes se beneficiaron del procesamiento de más de una muestra, por lo que sí que recomendamos la remisión de muestras de diferentes localizaciones en pacientes con EI dudosa o en los que se desconoce el agente causal en el momento de la cirugía.
La microbiología clásica es fundamental en el diagnóstico de la EI. La tinción de Gram destacó por su elevado VPP, 100% (IC95%, 74,7-100%); todos los pacientes en los que se observaron microorganismos en esta tinción presentaron EI. Además, se detectó una mayor sensibilidad de la PCR en aquellos casos en los que se observaron microorganismos, aunque no fue estadísticamente significativa como sí describen otros trabajos24,25, debido probablemente al tamaño muestral.
El hemocultivo fue la técnica que presentó una mayor sensibilidad de forma aislada y sigue siendo el referente en el diagnóstico microbiológico de la EI. El porcentaje de hemocultivos negativos fue del 13,5%, situado en la parte baja del rango recogido en la literatura (9-45%)3,26, y los microorganismos detectados por otras técnicas fueron cocos grampositivos y microorganismos intracelulares (tabla 4), coincidiendo con lo descrito en otros estudios3,25–27. Respecto a los pacientes con hemocultivo positivo y PCR negativa, destaca que todos habían llevado tratamiento antibiótico prolongado, lo que se asocia a una mayor proporción de PCR negativa24, aunque en nuestra serie no se detectaron diferencias significativas (tabla 4).
En nuestra serie, el cultivo valvular presentó un importante porcentaje de contaminaciones y no aportó ningún aislamiento que no se hubiera identificado por PCR universal, como se describe en publicaciones similares3,26,27. La sensibilidad de la PCR fue significativamente superior a la del cultivo (80,8 frente a 27,1%; p<0,001), lo que corroboran otros estudios26,29. En consecuencia, algunos autores abogan por priorizar las técnicas moleculares al cultivo o incluso sustituirlo26,27, aunque esto dejaría sin antibiograma a pacientes en los que el hemocultivo resulta negativo. Para subsanarlo se podría realizar un antibiograma molecular directamente de tejido valvular3, pero se detectaría un número limitado de resistencias.
La PCR universal contribuyó al diagnóstico etiológico en el 7,7% de los pacientes con EI definitiva. Aunque este porcentaje es importante, es inferior a los obtenidos en otros estudios como el de Peeters et al., en el que el 21% de los pacientes se benefició del resultado de la PCR 16S y en el 10% representó la única técnica microbiológica positiva29. En el trabajo de Shrestha et al. no se hubiera llegado al diagnóstico etiológico en el 14,4% de los pacientes sin la PCR 16S26, y en el de Mularoni et al., esta técnica fue la única positiva en un 41,6% de los pacientes3. La diferencia tan notable en cuanto al valor diagnóstico añadido se explica, además de por la menor sensibilidad de nuestra técnica, por el alto porcentaje de hemocultivos negativos en algunas series, como las de Shrestha et al.26 (25,1%) o Mularoni et al.3 (45%).
Cabe destacar que el diagnóstico microbiológico global (hemocultivo, PCR universal, PCR específica y serología) obtuvo unos elevados valores de sensibilidad, 96,2% (IC95%, 85,7-99,3), y especificidad, 98,2% (IC95%, 89-99,9), similares a los descritos en otros estudios3,27.
En nuestro estudio, el hemocultivo presentó una sensibilidad elevada y cabría plantearse el coste-efectividad de la PCR universal, pudiéndose obviar dicha prueba en pacientes con EI claramente filiada; sin embargo, en ocasiones microorganismos de la microbiota cutánea aislados en hemocultivos podrían considerarse responsables de la infección y enmascarar otro agente de crecimiento difícil. Asimismo, una PCR negativa ayudaría a descartar la endocarditis, como en el caso del paciente que presentamos con EI posible.
Por otro lado, los reactivos de la PCR universal son baratos, al tratarse de una técnica casera, siendo el tiempo de trabajo técnico lo más costoso. No obstante, el bajo número de muestras de pacientes con EI y su inclusión en la rutina no supone un coste que no esté justificado por los beneficios aportados, aun en un reducido número de pacientes.
Además, para crear un circuito robusto y evitar extraviar muestras valiosas, recomendamos remitir rutinariamente las muestras valvulares de quirófano a microbiología y anatomía patológica y, según el paciente, se realizarán pruebas adicionales o se conservará la muestra congelada, recuperándola en caso de mala evolución o discrepancias diagnósticas. La comunicación de las unidades implicadas a través del equipo de endocarditis es fundamental para que se lleve a cabo este procedimiento de forma eficiente.
Respecto al diagnóstico histopatológico, el 83,9% de los pacientes con EI definitiva presentó lesiones compatibles con endocarditis coincidiendo con otras series26,30. Sin embargo, la mayor sensibilidad del diagnóstico molecular en pacientes con histopatología positiva no fue estadísticamente significativa, al contrario que en otros trabajos24.
En relación con los 2 casos de EI posible, cabe señalar que, con la incorporación de las técnicas moleculares a los nuevos criterios de Duke de 2023, probablemente hubieran sido catalogadas como EI descartada o definitiva. En uno de estos pacientes, a pesar de presentar un aislamiento en el hemocultivo, la PCR universal resultó negativa, coincidiendo con la valoración macroscópica de la válvula en el momento de la cirugía y con los resultados negativos de la tinción de Gram y del cultivo. En este caso, la PCR nos permitiría descartar la EI y contradice a los autores que abogan por no realizar pruebas diagnósticas sobre la válvula en pacientes que presentan un aislamiento en hemocultivo31. En el otro caso de EI posible, en el que no se realizó hemocultivo ni examen histopatológico pero se observaron cocos grampositivos en la tinción de Gram y mediante PCR universal y cultivo valvular se identificó Streptococcus anginosus, destacamos la importancia de la visión de la tinción de Gram para corroborar los resultados del cultivo y de la PCR. Sin embargo, se han descrito algunos casos de pacientes con antecedentes de EI en los que la tinción de Gram y la PCR resultaron positivas, por lo que los resultados de estas técnicas deben interpretarse con cautela si no existe crecimiento en el cultivo valvular24,32.
ConclusionesEste trabajo aporta nuestra experiencia en el proceso de validación de la PCR universal, que se mostró como una técnica valiosa cuya sensibilidad fue significativamente superior a la del cultivo valvular. Completa el diagnóstico clásico, en el que destaca el hemocultivo, por ser la técnica microbiológica que presentó una mayor sensibilidad de forma aislada, y la tinción de Gram, por su elevado VPP. La mayor sensibilidad y especificidad se obtuvo al considerar conjuntamente la microbiología clásica y la molecular (fig. 3).
En caso de intervención, recomendamos siempre la remisión de muestras al servicio de microbiología y de anatomía patológica y su procesamiento o conservación hasta la resolución del episodio clínico. En una entidad tan compleja como la EI, es necesario utilizar todas las herramientas diagnósticas a nuestro alcance. Todas las áreas de diagnóstico de laboratorio son complementarias y no excluyentes, y deberían ser valoradas conjuntamente.
La importancia de las técnicas moleculares en el diagnóstico etiológico de la EI ha sido respaldada por su reciente inclusión en los criterios diagnósticos de Duke. Sin embargo, son técnicas no estandarizadas cuyo desarrollo es complejo y están restringidas a laboratorios de referencia, donde deben ser evaluadas y validadas antes de su instauración en la práctica clínica.
¿Qué novedades aporta?Aportamos la experiencia en el proceso de validación de la PCR universal, que se mostró como una técnica valiosa cuya sensibilidad fue significativamente superior a la del cultivo valvular (80,8 frente a 27,1%, p<0,0001) y añadió valor diagnóstico en el 7,7% de los pacientes con EI definitiva. También destacamos la importancia de las técnicas clásicas defendiendo la microbiología convencional frente a aquellas corrientes que abogan por su sustitución por técnicas moleculares.
Ninguna.
Consideraciones éticasEl estudio fue aprobado por el Comité de Ética del hospital (registro 2017/0271) y se obtuvo el consentimiento informado de los pacientes. No se detectaron diferencias por sexos.
Declaración sobre el uso de inteligencia artificialNo se ha empleado.
Contribución de los autoresP. Falomir Salcedo, J.M. Sahuquillo Arce, M. Blanes Juliá, M.D. Gómez Ruiz, A.M. Bel Mínguez, N. Mancheño Franch, M.Á. Arnau Vives, E. Ibáñez Martínez, J.L. López Hontangas y F.J. Chorro Gascó han contribuido a la concepción y el diseño, la adquisición de datos, su análisis e interpretación. P. Falomir Salcedo, J.M. Sahuquillo Arce, M. Blanes Juliá, M.D. Gómez Ruiz y F.J. Chorro Gascó han contribuido a la redacción del artículo o su revisión crítica. Todos los autores han dado su aprobación a la versión final del artículo.
Conflicto de interesesNinguno.